/home/virtual/kjpt/journal//../xmls/kjpt-45-2-145.xml
한국-일본과 중국-대만 석곡의 유전적 차이
적 요
우리나라 석곡과 석곡이 자연 분포하는 인접의 일본, 대만, 중국 석곡간의 유전적 차이를 규명하고 약재로 유통되는 석곡의 동종성을 검증하기 위하여, 4개국의 여러 집단에서 기원한 석곡 30여 개체 및 근연의 28종간 핵 ITS 지역 및 엽록체 rbcL, matK, trnH-psbA 지역 등의 염기서열을 비교분석하였다. 계통학적 분석 결과 한국산 석곡과 원기재지인 일본의 석곡은 단계통군을 이루었으며, 염기서열상으로도 큰 차이가 없어, 동일종으로 판단되었다. 그러나 중국 및 대만산 석곡은 한국 및 일본 석곡과는 매우 다른 분기군들을 이루어, 측계통군 또는 다계통군을 형성하였다. 근연의 다양한 석곡속 종들이 한국-일본 석곡과 중국-대만 석곡 사이에 위치하였고, 중국-대만 석곡 개체간에도 다양한 근연의 다른 종들이 위치하였다. 원래 석곡이 일본에서 기재되었으므로 한국-일본 석곡이 원종이다. 그러나 중국-대만 석곡은 다양한 다른 종의 복합체로 생각되며, 중국-대만 석곡의 경우 종의 실체 파악을 위하여 보다 자세한 조사가 필요하다. 우리나라-일본 석곡의 경우 집단 간의 유전적 차이가 거의 없었고, 대만-중국 석곡의 경우는 개체간의 유전적 이질성이 매우 높았다.
주요어: 석곡, 한국-일본산, 중국-대만산, 단계통군, 측계통군, 유전적 이질성
Abstract
The nucleotide sequences of nuclear ribosomal ITS regions and chloroplast rbcL, matK and psbA-trnH regions of 30 individuals of Dendrobium moniliforme from several localities in four countries and 28 related species of Dendrobium were compared to investigate the genetic differences among Korean, Japanese, Taiwanese and Chinese D. moniliforme, and to verify the homogeneity of D. moniliforme, which is used as a traditional medicine in East Asia. A phylogenetic analysis showed that Korean D. moniliforme and Japanese D. moniliforme form a monophyletic group, with no significant differences between their nucleotide sequences. This confirms that they are the same species. However, the Chinese and Taiwanese D. moniliforme were polyphyletic. Various species related to D. moniliforme were located between the Korean-Japanese D. moniliforme and the Chinese-Taiwanese D. moniliforme, and other related species were found between individuals of Chinese-Taiwanese D. moniliforme. D. moniliforme is described in Japan, providing evidence that the Korean-Japanese D. moniliforme is the original species. In addition, our data suggest that the Chinese-Taiwanese D. moniliforme complex is a mixture of a range of other species. Further studies are required to understand the taxonomic identity of this species. In the Korean-Japanese D. moniliforme, there were almost no genetic differences among the localities, whereas the genetic heterogeneity was high among individuals of the Chinese-Taiwanese D. moniliforme.
Keywords: Dendrobium moniliforme, Korean-Japanese origin, Chinese-Taiwanese origin, monophyletic, paraphyletic, genetic heterogeneity
석곡속( Dendrobium Swartz)은 석곡( Dendrobium moniliforme(L.) Swartz) 외 1000여 종으로 구성된 큰 속이다. 석곡은 한국, 중국, 일본, 대만에 분포한다. 석곡은 일본에서 채집된 개체를 관찰한 린네에 의해 Epidendrum moniliforme L. (1753)으로 처음 명명되었다. 이후 Swartz가 새로운 속인 Dendrobium속을 만들면서 Dendrobium moniliforme (L.) Swartz (1799)로 이전하였다. 석곡속은 한국, 일본에서부터 동남아시아, 파푸아뉴기니, 오스트레일리아에 이르기까지 세계적으로 널리 분포한다( Lee, 2007). 국내에서 석곡은 멸종위험도가 높은 종 중의 하나로, 환경부는 석곡을 멸종위기 야생동식물 2급으로 지정하여 보호하고 있다. 우리나라 남부에서 자생지가 몇 곳 발견되어 개체들이 보호받고 있으나, 집단이 작게 파편화되어있고 자생 개체수가 매우 적다. 전라남도, 경상남도, 제주도 그리고 일부 도서지역에 분포하는 것으로 알려져 있다. 빛이 비추는 곳의 나무 표면이나 바위 겉에 착생하며 총상꽃차례 끝에 1-3개의 백색내지 연분홍색 꽃을 6-8월에 피운다.
석곡의 자생지와 개체 수는 한정적이지만, 일부 석곡개체가 전라남도나 경상남도의 섬과 산의 절벽처럼 사람의 접근이 힘든 곳에서 가끔 발견되고 있다. 시장에서 관상용으로 유통되는 석곡의 대부분은 위험에 처하기 이전 세대에 채취된 것들이 대량증식된 것이거나 수입된 것들이다. 그러나 재배개체들의 기원을 명확하게 규명하기는 어려우며, 많은 수의 재배개체들은 일본이나 중국으로부터 유입되어 대량증식된 것으로 추측된다.
따라서 본 연구는 국내 석곡 개체를 중국과 일본의 개체들과 구분할 수 있는 마커를 찾기 위하여 수행하였다. 먼저 이 논문에서는 핵과 엽록체 유전체에서 4개 DNA 마커의 염기서열을 생산하여 국내 개체, 국외 개체들의 유전적 차이를 계산한 후, 근연종을 포함한 계통수를 제작하여 국내외 석곡 개체 간에 어떤 유전적 차이점이 있는지 비교분석하였다.
재료 및 방법
재료
본 연구는 D. moniliforme 30개체와 근연종을 대표하는 28개체를 대상으로 수행하였다. D. moniliforme 의 경우 14개체는 국내 자생 개체로부터 직접 잎을 채취한 후 DNA를 추출하여 염기서열을 결정하였다. 이를 제외한 D. moniliforme 16개체에 대한 염기서열과 근연종에 포함되는 28개체의 염기서열은 GenBank에서 내려받아 계통분석에 사용하였다( Table 1). 직접 재료를 수집한 D. moniliforme 14개체는 우리나라 남쪽 해안가 및 내륙에 자생하는 자생지로부터 수집되었거나, 이들 지역에서 채집하여 고려대학교 연구온실 및 바보난농원 연구온실 등에 이식하여 재식하고 있는 개체로부터 채집하였는데, 그 기원지는 해남(3개체), 진도(5개체), 완도(1개체), 제주도(1개체), 보길도(1개체), 월출산(2개체), 갈곶도(1개체) 등이다( Table 1). 야생에서 채취한 개체들은 환경부의 멸종위기종 채취허가를 취득하여 채취하였고, 이식한 개체들도 멸종위기종 증식 복원을 위하여 허가된 개체들만 이용하였다. 채취한 잎은 아이스박스에 냉매와 함께 넣어 실험실로 이동한 후, -70oC 냉동고에 2-3일간 보관한 다음 DNA 추출에 사용하였다.
DNA 추출, PCR, 정제 및 염기서열결정 방법
DNA는 DNeasy plant mini kit(Qiagen, Germany)를 사용하여 추출하였다. 추출한 DNA의 증폭은 ITS primer 2쌍( White et al., 1990; Sun et al., 1994), rbcL primer 1쌍( Fay et al., 1997), matK primer 1쌍( CBOL Plant Workng Group, 2009), psbA- trnH 1쌍( Sang et al., 1997)을 이용해 수행하였다. PCR 산물은 PCR 정제 키트(iNtRON Biotechnology)를 이용하여 정제하였고, 정제된 PCR 산물은 ABI3730xl DNA sequencer로 염기서열을 결정하였다(Applied Biosystems).
계통분석
양쪽 방향으로 얻어진 염기서열은 Geneious Pro 6.1.8(Biomatters Ltd.)을 이용하여 정리하였다. 본 연구에서 얻어진 석곡 14개체의 nrITS, matK, rbcL, psbA- trnH지역의 염기서열 정보와 GenBank에서 얻은 염기서열을 Clustal W ( Thompson, 1994)를 통해 정렬하였다. 각 DNA barcode 지역과 산지별로 유전적 거리를 계산하기 위하여 MEGA6 ( Tamura, 2013)을 이용하여 Kimura-2-parameter(K2P)( Kimura, 1980) 방법을 적용하였다. PAUP 4.0b10( Swofford, 2002)을 이용하여 Neighbour Joining (NJ), Maximum Likelihood (ML) 분석을 수행하였고 NJ, ML 계통수에서 부트스트랩 값은 100회 반복을 통하여 산출하였다. ML분석을 위한 염기치환 모델은 Modeltest 3.7( Posada and Crandall, 1998)을 통해 nrITS지역은 GTR + G 모델을, 엽록체 3개 지역의 통합 자료는 F81 + G + I 모델을 선정하였다. Dendrobium 절과 근연인 Densiflora 절의 D. densiflora를 외군으로 이용하여 계통수의 뿌리를 결정하였다.
결 과
이 연구를 통하여 4개 DNA 마커 지역에 대하여 총 84개(직접 생산한 개체 14개체 × 6개 마커 지역, nrITS, matK, rbcL, psbA-trnH, atpF-H, psbK-I)의 염기서열이 얻어졌다. 그러나 atpF-H 와 psbK-I의 경우는 비교 대상이 되는 다른 개체 및 종들에서 이용이 불가능하여 분석에서 제외하였다. nrITS 지역의 염기서열 길이는 633 bp이며 GC%는 52.6%로 나타났다. 633 bp 중 133개 위치에서 변이를 보였는데, 이 중 19개는 1−3 bp indel 이었으며 나머지는 모두 염기치환에 의한 변이로 밝혀졌다. 114개의 염기치환변이 중 36개는 공유파생형질이고 78개가 단독파생형질이었다. 한국의 14개 accession은 indel 없이 1개의 단독파생형질과 1개의 공유파생형질로 총 2개의 위치에서 변이를 보였다.
엽록체 matK, rbcL 지역의 염기서열 길이와 GC%는 각각 794 bp, 32.2%와 623 bp, 42.7%로 나타났다. 엽록체 matK 지역은 794 bp 중 4개 위치에서 변이를 보였으며 모두 공유파생형질이었다. 엽록체 rbcL 지역은 623 bp 중 일본에서 보고한 서열의 1개 위치(350번 위치)에서 염기치환변이를, 한국산 1개 서열(734번 위치)이 1개의 indel을 가진 것으로 나타났다. 한국의 다른 13개 accession은 두 지역 모두에서 염기서열의 변화가 없는 것으로 확인되었다.
엽록체 IGS psbA- trnH 지역의 염기서열은 각각 601 bp의 길이와 35.6%의 GC%를 가진 것으로 나타났다. psbA- trnH 지역은 601 bp 중 5개 위치에서 공유파생형질, 4개 위치에서 단독파생형질의 염기치환변이가 관찰되었다. psbA- trnH 지역은 국내 4개 accession(재배종, 월출산, 해남, 진도)에서 변이가 있었다( Table 2). 엽록체 유전체 마커 3가지를 유합분석할 때 석곡 3개 개체들에 대하여는 2개의 마커만 가용할 수 있어서, 세 번째 마커는 결여형질로 처리하였다. 한국산 석곡과 주변국 석곡의 계통학적 위치를 확인하기 위해 Dendrobium 속 내의 Dendrobium 절의 15종 57 accession과 이들의 외군인 Densiflora 절의 D. densiflora 1종을 포함하여 총 58개의 nrITS 염기서열을 이용한 계통수를 제작하였다. Maximum Likelihood(ML) 최대값은 3219.97이었다. Neighbor Joining (NJ), ML, 부트스트랩 값 (BP)이 50% 이상인 clade에 한하여 NJ, ML 계통수에 표시하였다. nrITS 지역의 NJ, ML 분석결과 한국과 일본에서 보고된 석곡(한국-일본 type)은 단계통군을 형성하였다. 반면 중국과 대만에서 보고된 석곡은 Dendrobium 절 안의 다른 종들과 섞여 측계통군을 형성하였다. 한국-일본 type들은 NJ, ML BP가 각 93%, 91%로 단계통을 형성하였다( Fig. 1, Fig. 2). 엽록체 matK, rbcL, psbA- trnH 지역의 염기서열을 유합한 계통수는 Dendrobium 절의 12종 37 accession과 Densiflora 절의 1종 1 accession, 총 12종 38 accession을 이용하여 작성하였다. ML 최대값은 2657.64이었다. NJ, ML BP가 50% 이상인 clade에 한하여 NJ, ML 계통수에 표시하였다. NJ, ML분석 결과 한국-일본 type은 두 계통수 모두에서 66%의 부트스트랩 값으로 단계통군을 형성하였다. 함께 분석된 중국의 3개체 (China 1: KF143699 ( matK), KF177630 ( rbcL), KF177530 ( psbA- trnH); China 2: KF143700, KF177631, EU887938; China 3: KF361618, KF177632, GQ162803)는 다른 종들과 섞여 단계통군을 형성하지 아니하였다( Fig. 3, Fig. 4). 직접 생산한 국내 14개 개체의 DNA 염기서열과 NCBI에 보고된 국내 개체들에 대하여 측정한 유전적 거리는 psbA- trnH 지역이 0.00131 ± 0.00164 (Mean ± S.D.) 로 가장 높게 나타났으며 rbcL 지역이 0.00018 ± 0.00051 로 가장 낮게 나타났다. 나머지 지역(nrITS, matK)의 유전적 거리는 각각 0.00107 ± 0.00096, 0.0014 ± 0.00255 이었다( Table 3). 한국이 아닌 다른 국가로부터 NCBI에 보고된 서열들을 대상으로 유전적 거리를 구하였는데, nrITS 지역의 유전 적 거리는 0.04122 ± 0.0435로 가장 높았으며 rbcL 지역이 0.00046 ± 0.00074로 가장 낮은 값을 보였다. 나머지 2개 지역(matK, psbA-trnH)의 값은 각각 0.00132 ± 0.00094, 0.00145 ± 0.00107이었다.
직접 생산한 서열과 NCBI로부터 획득한 서열 모두를 대상으로 계산한 유전적 거리는 nrITS 지역이 0.02759 ± 0.03629로 가장 높았고, rbcL 지역이 0.00014 ± 0.00045로 나타났다. matK 지역과 psbA-trnH 지역의 거리는 0.00101 ± 0.00126, 0.00241 ± 0.00301이었다. 한국과 일본에서 보고된 서열만을 대상으로 계산한 유전적 거리는 nrITS 지역이 0.00184 ± 0.0023, matK 지역은 0.00125 ± 0.00236, rbcL 지역은 0.00034 ± 0.00068 이었다. psbA-trnH 지역은 일본 지역에서 보고된 서열이 없어 계산하지 아니하였다.
고 찰
이 연구를 통해 생산된 14개 국내 석곡 개체들에 대해 K2P 거리를 이용한 유전적 다양성 검증을 실시한 결과, 4개 마커 전부에 대하여 국내 개체들의 유전적 다양성이 낮다는 결과를 얻었다( Table 3). Infat et al. (2012)는 Paphiopedilum 7-8개 종에 대하여 0-0.0016의 K2P 유전적 거리 값을 보고하였다. 이 값은 국내 개체들 간에 계산한 K2P 유전적 거리 값과 비슷하다. Tsai et al. (2004)은 타이완에 서식하는 Dendrobium속을 대상으로 ITS 지역의 유전적 거리 값을 계산하였다. 그 결과, 가장 낮게 보고된 유전적 거리 값은 국내 개체들의 유전적 거리 값에 비해 60배 가까이 높았다. 국내 D. moniliforme가 낮은 유전적 다양성을 보이는 이유는 두 가지로 추측할 수 있다. 첫 번째로, 국내의 석곡이 과거 지사학적으로 오랜 시간에 걸쳐 집단의 수축과 팽창과 같은 병목현상을 겪어 유전적 이질성이 낮아졌을 가능성을 들 수 있다. 이는 외국에서 보고된 염기서열들 간의 유전적 거리 값이 국내 개체들 간의 값과 비슷하거나 40배에 가깝게 높은 것으로 확인할 수 있다( Table 3). 두 번째로, 난과 식물이 다른 분류군에 비하여 4개 DNA barcode 지역 서열의 유전적 다양성이 낮을 가능성이다. 여러 분류군을 대상으로 유전적 거리 값을 계산한 결과들이 보고되어 있다( Chen et al., 2010; Shi et al., 2011; Zhang et al., 2012, Kim et al., 2014). 엽록체 지역 마커에 대하여는 유전적 거리 값의 변이가 커서 단정할 수 없지만, nrITS 지역에 대해서는 위에 언급된 논문에 사용된 분류군들과 비교하여 보았을 때 난과 식물의 유전적 거리값이 낮았다. 본 연구의 결과에서 가장 주목할 점으로는 우리나라 석곡( D. moniliforme)과 일본 석곡 개체들은 nrITS 및 엽록체 3개 마커를 유합한 계통수에서 모두 단계통군을 형성하였다는 것을 들 수 있다. 한편, 중국과 대만 개체들은 계통수에서 근연종들과 섞여 측계통군을 형성하였다( Fig. 1, Fig. 2, Fig. 3, Fig. 4). 특히 근연종이 많이 이용된 nrITS 계통수에서 한국-일본 type의 석곡은 ML 및 NJ 계통수에서 모두 90% 이상의 BP로 지지되는 단계통군을 이룬 반면, 중국-대만산 석곡의 경우 최소한 3개 이상의 그룹에 흩어져 위치하는 것으로 확인되었다. 이중 한국-일본산 석곡에 가장 가까운 중국-대만산 석곡 그룹에는 D. okinawense 및 D. henanense 등 다른 종들이 위치한다. 두 번째 대만-중국산 석곡 그룹에도 D. wilsonii, D. housshanense, D. fanjingshanense 등이 위치한다. 여기에 언급된 5종은 모두 일본 및 중국 특산종으로써 국지적으로 분포하는 종들인데, 이들 종의 형태적 및 분자적 특성이 조사되어야만 한국-일본산 석곡과의 유연관계가 정확하게 설명될 수 있을 것이다. 종 수에서 제한적이기는 하지만 엽록체 유전체의 3개 마커를 이용한 계통수에서도 같은 근연종들이 한국-일본 및 중국, 대만 석곡들 사이에 위치하였다( Fig. 3, Fig. 4). 석곡의 종의 범위를 위에 언급한 5개의 종을 모두 포함하도록 넓게 인지하더라도 계통수의 하단부에 존재하는 또 다른 중국-대만 석곡이 존재하므로 현재 인지하는 한국-일본, 대만, 중국 석곡이 최소한 측계통군인 것은 명확하다. 본 연구의 분석에 사용된 석곡 개체들의 염기서열을 따로 정렬하였을 때, nrITS의 133개 위치에서 변이를 발견할 수 있었다( Fig. 5). 이 중 10개 위치에서 원산지별로 뚜렷이 구분되는 염기치환변이를 확인할 수 있었다. 한국 기원 14개체, 일본 기원 2개체가 중국 기원 10개체, 대만 기원 4개체와 특히 잘 구분되는 위치는 127번 위치이다. 이 외에도 57번, 487번, 600번 위치들도 서열이 보고된 지역에 따라 구분될 정도의 염기치환변이를 보였다. 이 차이를 통해 제한된 시료에 바탕을 둔 것이긴 하지만 한국-일본 type의 석곡과 중국-대만 type의 석곡을 구분하는 것이 가능하다. Ding et al.(2008)은 기원지에 따라 D. officinale Kimura et Migo의 nrITS 염기서열에 차이가 있다고 보고하였는데, 이는 같은 속에 속하는 석곡( D. moniliforme)이 기원지에 따라 염기서열의 변화를 보일 가능성이 있음을 암시한다. 한국과 일본의 석곡도 정렬된 염기서열 자료에서 몇 개의 갭(gap)으로 구분할 수 있었으나, 일본 기원 개체의 수가 2개로 매우 적고, 보고된 서열의 갭이 많아 염기서열 판독에 오류가 있을 가능성을 생각하여 한국과 일본 개체를 구분하기 위해서는 추가적인 개체의 확보와 실험이 필요하다고 판단된다. 대만에서 보고된 개체들의 경우 그 중 하나가 외군과 가까이 계통학적 위치가 형성되는 것을 확인할 수 있는데, 염기서열의 판독이 잘못되었거나 사용된 표본이 오동정되었을 가능성이 있다. 그러나 중국-대만 개체들도 유집되는 경향성을 보이고 있기에 기존에 출판된 자료들이 모두 오동정이라고 보기에는 무리이다. 따라서 중국-대만 개체들이 측계통군 내지 다계통군을 형성한 것에는 종의 인지범위 차이, 오동정 등의 분류학적 문제가 복합적으로 작용했을 가능성이 있다. 엽록체 3개 마커를 이용해 얻어진 염기서열을 정렬하여 얻은 두 계통수(NJ, ML) 역시 nrITS에서 얻어진 결과를 지지한다. 한국-일본 개체들은 NJ 계통수에서 58%의 부트스트랩 값, ML 계통수에서 66%의 부트스트랩 값으로 지지되는 단계통군을 형성하였다. 같이 비교된 중국 3개 accession은 근연종들과 섞여 측계통군을 이루는 것을 NJ 및 ML 계통수 모두에서 확인하였다. 염기서열을 정렬하여 보았을 때, matK와 rbcL의 경우 각 794 bp, 623 bp에서 변이가 일어난 위치 개수가 각각 4개와 2개로 매우 적었는데, 이는 이들 유전자의 진화 속도가 nrITS에 비해 느리기 때문인 것으로 추측된다. matK 유전자의 경우 다양한 식물군에서 진화 속도가 비교된 적이 있으며(Hollinigsworth et al., 2011; Fazekas et al., 2012), rbcL의 염기서열은 그 진화 속도가 식물 종별로 다양하다는 것이 알려진 적이 있다(Bousquet et al., 1992; Asahina et al., 2010). nrITS와는 다르게 염기서열상에서 지역별로 특별히 구분되는 위치는 찾을 수 없었다. 그럼에도 엽록체 3개 마커를 이용한 계통수는 한국 개체들과 중국 개체들을 구분하고 있었다. 린네는 1753년 일본에서 채집된 개체를 이용하여 석곡을 처음 기재하였다. nrITS 마커를 이용한 모든 계통수에서 한국-일본 type이 단계통군을 형성하는 것과 염기서열의 동일성을 보았을 때, 한국-일본 type이 처음 기재된 같은 원종임을 확인할 수 있다. 엽록체 DNA를 이용한 모든 계통수 제작 결과 또한 한국 및 일본 개체들이 단계통을 형성함으로써 이들의 동종성을 지지하였다. 그러나 중국-대만 type의 석곡은 최소한 3개 종 이상으로 구성된 다양한 종들의 복합적인 이름으로 생각되며, 실체 파악을 위하여 Dendrobium절 내의 다른 종들과 상세히 비교하는 보다 폭넓은 형태적 및 분자적 조사가 필요하다. 제한적이긴 하지만 우리나라 석곡은 일본의 석곡과 함께 기본종이고 중국원산의 석곡은 우리나라 석곡과는 크게 차이가 나므로, 중국 원산의 석곡을 번식하여 국내로 도입된 개체들을 본 연구자들이 이용한 마커들을 이용하여 대부분 구별할 수 있을 것이다. 또한 널리 재배하는 D. moniliforme는 다양한 종간잡종에서 기원한 품종들이 포함되므로, 재배품종들의 명확한 기원을 규명하기 위해서는 광범위한 자생개체 및 재배개체들에 대한 유전적 비교연구가 추가되어야 할 것이다.
ACKNOWLEDGMENTS
본 연구는 환경부 “차세대 에코이노베이션 기술개발사업(과제번호 416-111-007)”으로 지원받은 과제이다. 사용된 DNA sample은 한국의 식물 DNA 은행(2013-1829, 2013-1830, 2013-1831, 2013-1832, 2013-1833, 2013-1834, 2013-1835, 2013-1836, 2013-1837, 2014-1826, 2014-1827, 2014-1828, 2014-1829, 2014-1830)에 보관되고 있으며 분양가능하다.
Fig 1.
Neighbour Joining tree of Dendoribum based on the nuclear ribosomal ITS region. Phylogenetic position of Dendoribum moniliforme was evaluated in this study. NJ bootstrap support values more than 50% are given on the above or on the below each branch. 
Fig 2.
Maximum Likehood (ML) tree of Dendoribum based on nuclear ribosomal ITS region. Phylogenetic position of Dendoribum moniliforme was evaluated in this study. ML bootstrap support values more than 50% are given on the above or on the below each branch. 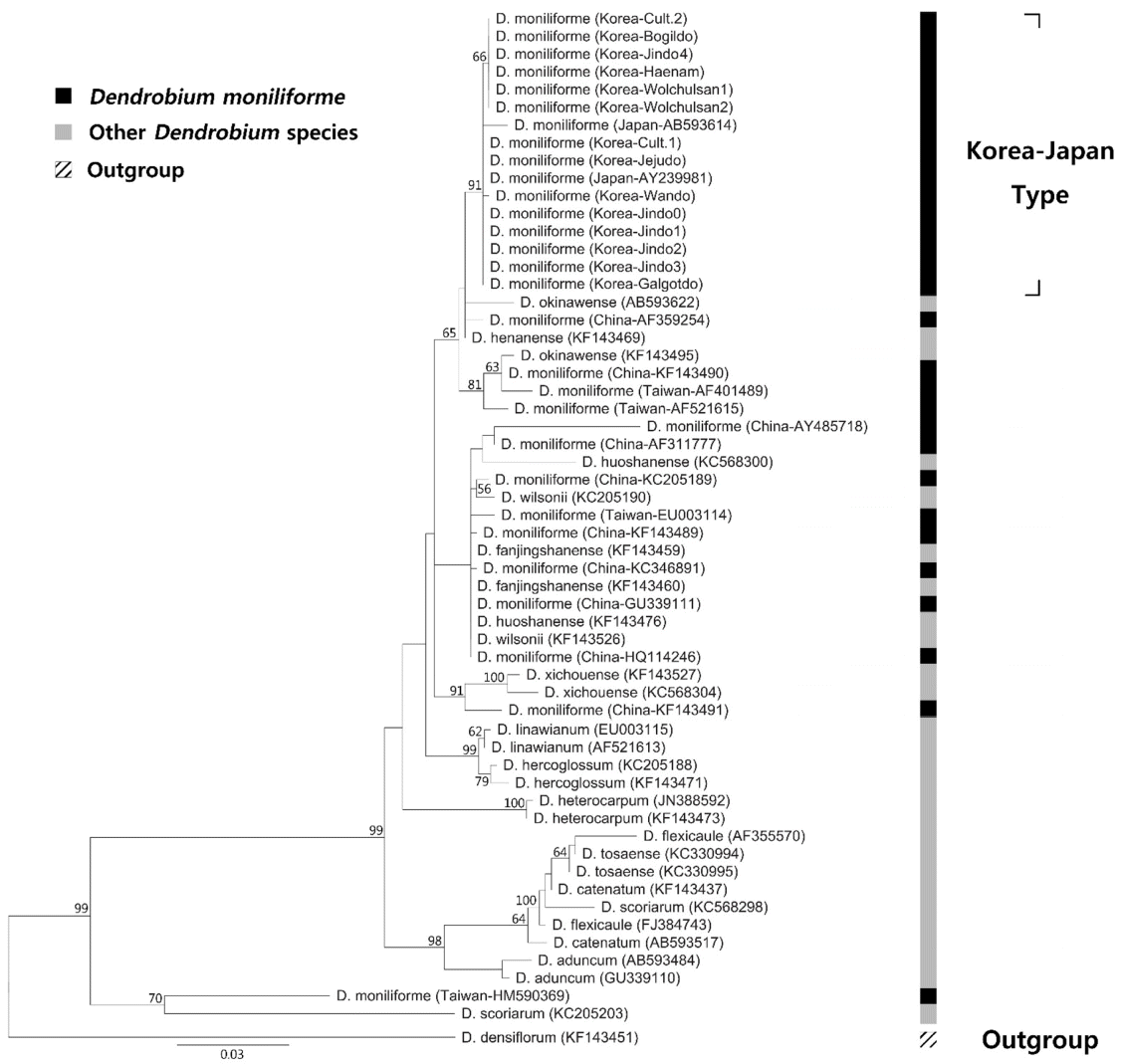
Fig 3.
Neighbour Joining tree of Dendoribum based on the three chloroplast DNA markers (rbcL, matK, and psbA-trnH) regions. Phylogenetic position of Dendoribum moniliforme was evaluated in this study. NJ bootstrap support values more than 50% are given on the above or on the below each branch. 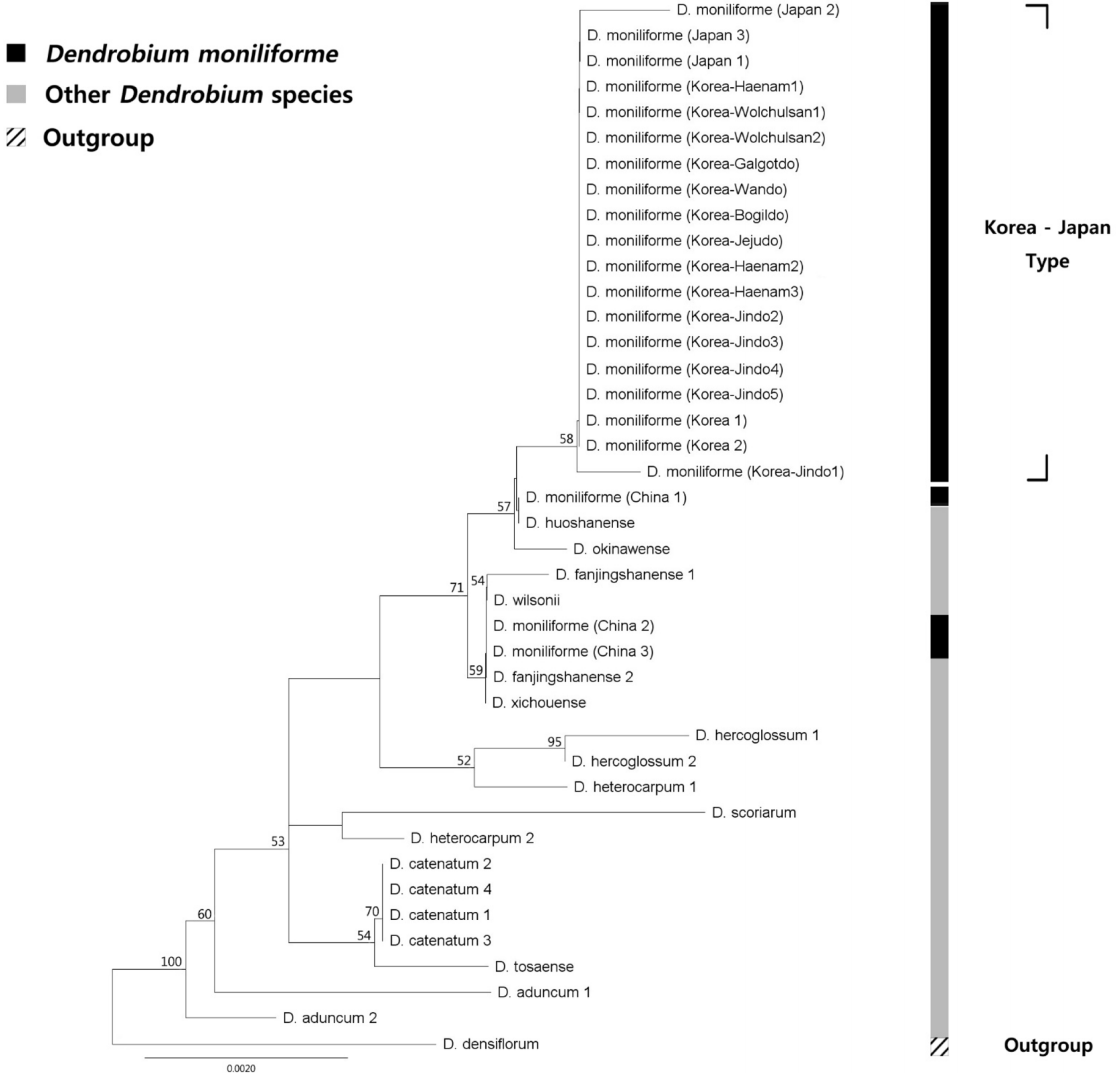
Fig 4.
Maximum Likehood (ML) tree of Dendoribum based on he three chloroplast DNA markers (rbcL, matK, and psbA-trnH) regions. Phylogenetic position of Dendoribum moniliforme was evaluated in this study. ML bootstrap support values more than 50% are given on the above or on the below each branch. 
Fig 5.
The sequence variation of nuclear ribosomal ITS region among 30 Dendoribum moniliforme accessions. 
Table 1.
Sample data used in this study (Sample accessions with asterisk are new DNA sequences generated in this study.).
|
Species |
nrITS |
matK |
rbcL |
psbA-trnH |
Origin |
|
*D. moniliforme (L.) Sw. |
KP686385 |
KP686315 |
KP686357 |
KP686329 |
Jindo 1 |
|
*D. moniliforme
|
KP686377 |
KP686307 |
KP686349 |
KP686321 |
Jindo 2 |
|
*D. moniliforme
|
KP686378 |
KP686308 |
KP686350 |
KP686322 |
Jindo 3 |
|
*D. moniliforme
|
KP686379 |
KP686309 |
KP686351 |
KP686323 |
Jindo 4 |
|
*D. moniliforme
|
KP686381 |
KP686311 |
KP686353 |
KP686325 |
Jindo 5 |
|
*D. moniliforme
|
KP686380 |
KP686310 |
KP686352 |
KP686324 |
Haenam 1 |
|
*D. moniliforme
|
KP686383 |
KP686313 |
KP686355 |
KP686327 |
Haenam 2 |
|
*D. moniliforme
|
KP686384 |
KP686314 |
KP686356 |
KP686328 |
Haenam 3 |
|
*D. moniliforme
|
KP686382 |
KP686312 |
KP686354 |
KP686326 |
Wando |
|
*D. moniliforme
|
KP686386 |
KP686316 |
KP686358 |
KP686330 |
Jejudo |
|
*D. moniliforme
|
KP686387 |
KP686317 |
KP686359 |
KP686331 |
Bogildo |
|
*D. moniliforme
|
KP686388 |
KP686318 |
KP686360 |
KP686332 |
Wolchulsan 1 |
|
*D. moniliforme
|
KP686389 |
KP686319 |
KP686361 |
KP686333 |
Wolchulsan 2 |
|
*D. moniliforme
|
KP686390 |
KP686320 |
KP686362 |
KP686334 |
Galgotdo |
|
D. aduncum Lindl. |
- |
- |
- |
FJ216483 |
China |
|
D. aduncum
|
- |
KF143427 |
KF177571 |
KF177467 |
China |
|
D. aduncum
|
GU339110 |
- |
- |
- |
China |
|
D. aduncum
|
- |
HM055147 |
JF713125 |
- |
India |
|
D. aduncum
|
AB593484 |
- |
- |
- |
Japan |
|
D. catenatum Lindl. |
- |
- |
KF177582 |
KF177478 |
China |
|
D. catenatum
|
- |
- |
KF177580 |
KF177476 |
China |
|
D. catenatum
|
- |
- |
KF177581 |
KF177477 |
China |
|
D. catenatum
|
- |
- |
KF177583 |
KF177479 |
China |
|
D. catenatum
|
- |
KF143649 |
- |
- |
China |
|
D. catenatum
|
KF143437 |
KF143647 |
- |
- |
China |
|
D. catenatum
|
- |
KF143648 |
- |
- |
China |
|
D. catenatum
|
- |
KF143650 |
- |
- |
China |
|
D. catenatum
|
AB593517 |
- |
- |
- |
Japan |
|
D. densiflorum
|
KF143451 |
KF143661 |
KF177594 |
KF177490 |
China |
|
D. fanjingshanense Z.H.Tsi ex X.H.Jin & Y.W.Zhang |
- |
- |
KF177602 |
KF177499 |
China |
|
D. fanjingshanense
|
KF143459 |
KF143669 |
KF177601 |
KF177498 |
China |
|
D. fanjingshanense
|
KF143460 |
KF143670 |
- |
- |
China |
|
D. flexicaule Z.H.Tsi, S.C.Sun & L.G.Xu |
FJ384743 |
- |
- |
- |
China |
|
D. flexicaule
|
AF355570 |
- |
- |
- |
China |
|
D. henanense J.L.Lu & L.X.Gao |
KF143469 |
- |
- |
- |
China |
|
D. hercoglossum Rchb.f. |
KF143471 |
KF143681 |
KF177612 |
KF177510 |
China |
|
D. hercoglossum
|
- |
- |
KF177613 |
KF177511 |
China |
|
D. hercoglossum
|
- |
KF143682 |
- |
- |
China |
|
D. hercoglossum
|
KC205188 |
- |
- |
- |
China |
|
D. heterocarpum Wall. ex Lindl. |
JN388592 |
- |
- |
- |
China |
|
D. heterocarpum
|
- |
- |
FJ216556 |
FJ216471 |
China |
|
D. heterocarpum
|
KF143473 |
KF143683 |
KF177614 |
KF177512 |
China |
|
D. heterocarpum
|
- |
AB847779 |
- |
- |
Japan |
|
D. huoshanense Z.Z.Tang & S.J. Cheng |
KF143476 |
KF143686 |
KF177617 |
KF177515 |
China |
|
D. huoshanense
|
KC568300 |
- |
- |
- |
China |
|
D. linawianum Rchb.f. |
EU003115 |
- |
- |
- |
Taiwan |
|
D. linawianum
|
AF521613 |
- |
- |
- |
China |
|
D. moniliforme
|
- |
- |
FJ216568 |
EU887938 |
China |
|
D. moniliforme
|
KF143489 |
KF143698 |
KF177630 |
KF177528 |
China |
|
D. moniliforme
|
AF521615 |
- |
- |
- |
China |
|
D. moniliforme
|
- |
- |
- |
EU672796 |
China |
|
D. moniliforme
|
- |
- |
- |
GQ162803 |
China |
|
D. moniliforme
|
- |
KF361618 |
- |
- |
China |
|
D. moniliforme
|
AF311777 |
- |
- |
- |
China |
|
D. moniliforme
|
KF143491 |
KF143700 |
KF177632 |
KF177530 |
China |
|
D. moniliforme
|
KF143490 |
KF143699 |
KF177631 |
KF177529 |
China |
|
D. moniliforme
|
HQ114246 |
- |
- |
- |
China |
|
D. moniliforme
|
KC205189 |
- |
- |
- |
China |
|
D. moniliforme
|
KC346891 |
- |
- |
- |
China |
|
D. moniliforme
|
GU339111 |
- |
- |
- |
China |
|
D. moniliforme
|
AF359254 |
- |
- |
- |
China |
|
D. moniliforme
|
AY485718 |
- |
- |
- |
China |
|
D. moniliforme
|
AY239981 |
- |
- |
- |
Japan |
|
D. moniliforme
|
- |
AB519774 |
AB519787 |
- |
Japan |
|
D. moniliforme
|
- |
AB519773 |
AB519788 |
- |
Japan |
|
D. moniliforme
|
- |
AB519775 |
AB519786 |
- |
Japan |
|
D. moniliforme
|
AB593614 |
AB847816 |
- |
- |
Japan |
|
D. moniliforme
|
- |
- |
- |
KF262193 |
Korea |
|
D. moniliforme
|
- |
KC704627 |
KC704892 |
KC704360 |
Korea |
|
D. moniliforme
|
- |
KC704628 |
KC704893 |
KC704361 |
Korea |
|
D. moniliforme
|
EU003114 |
- |
- |
- |
Taiwan |
|
D. moniliforme
|
HM590369 |
- |
- |
- |
Taiwan |
|
D. moniliforme
|
AF401489 |
- |
- |
- |
Taiwan |
|
D. okinawense Hatus. & Ida |
KF143495 |
KF143704 |
KF177636 |
KF177534 |
China |
|
D. okinawense
|
AB593622 |
- |
- |
- |
Japan |
|
D. scoriarum W.W.Sm. |
- |
KF143717 |
KF177649 |
KF177546 |
China |
|
D. scoriarum
|
KC568298 |
- |
- |
- |
China |
|
D. scoriarum
|
KC205203 |
- |
- |
- |
China |
|
D. tosaense Makino |
KC330994 |
- |
- |
- |
China |
|
D. tosaense
|
KC330995 |
- |
- |
- |
China |
|
D. tosaense
|
- |
- |
- |
EU672794 |
China |
|
D. tosaense
|
- |
AB519771 |
- |
- |
Japan |
|
D. tosaense
|
- |
- |
AB519782 |
- |
Japan |
|
D. wilsonii Rolfe |
KF143526 |
KF143735 |
KF177666 |
KF177563 |
China |
|
D. wilsonii
|
KC205190 |
- |
- |
- |
China |
|
D. xichouense S.J.Cheng & Z.Z.Tang |
- |
- |
KF177667 |
KF177564 |
China |
|
D. xichouense
|
KF143527 |
KF143736 |
- |
- |
China |
|
D. xichouense
|
KC568304 |
- |
- |
- |
China |
Table 2.
Statistic data for four DNA markers (nrITS, rbcL, matK, psbA-trnH) used in the comparison of Dendrobium moniliforme.
|
nrITS |
matK |
rbcL |
psbA-trnH |
|
Aligned length(bp) |
633 |
794 |
623 |
601 |
|
% GC |
52.60 |
32.20 |
42.70 |
35.60 |
|
Number of constant characters |
500 |
790 |
621 |
592 |
|
Number of parsimony-uninformative characters |
101 |
0 |
2 |
4 |
|
Number of parsimony-informative characters |
32 |
4 |
0 |
5 |
|
Pairwise identity (%) |
97.10 |
99.90 |
99.90 |
99.70 |
Table 3.
The K2P distance of among different accessions of Dendrobium moniliforme.
|
nrITS |
matK |
rbcL |
psbA-trnH |
|
Number of Taxa |
30 |
23 |
23 |
23 |
|
K2Ps among domesitc accessions |
0.00107 (± 0.00096) |
0.0014 (± 0.00255) |
0.00018 (± 0.00051) |
0.00131 (± 0.00164) |
|
K2Ps among foreign accessions |
0.04122 (± 0.0435) |
0.00132 (± 0.00094) |
0.00046 (± 0.00074) |
0.00145 (± 0.00107) |
|
K2Ps among Korean-Japanese accessions |
0.00184 (± 0.00230) |
0.00125 (± 0.00236) |
0.00034 (± 0.00068) |
NA*
|
|
K2Ps among entire accessions |
0.02759 (± 0.03629) |
0.00101 (± 0.00126) |
0.00014 (± 0.00045) |
0.00241 (± 0.00301) |
Literature Cited
Arca, M. Hinsinger, DD. Cruaud, C. Tillier, A. Bousquet, J and Frascaria-Lacoste, N. 2012. Deciduous trees and the application of universal DNA barcodes: a case study on the circumpolar Fraxinus. Plos One 7(3): e34089.  Asahina, H. Shinozaki, J. Masuda, K. Morimitsu, Y and Satake, M. 2010. Identification of medicinal Dendrobium species by phylogenetic analyses using matK and rbcL sequences. Journal of Natural Medicines 64: 133-138.   Barrett, CF and Freudenstein, JV. 2008. Molecular evolution of rbcL in the mycoheterotrophic coralroot orchids ( Corallorhiza Gagnebin, Orchidaceae). Molecular Phylogenetics and Evolution 47: 665-679.   Cai, X. Feng, Z. Zhang, X. Xu, W. Hou, B and Ding, X. 2011. Genetic diversity and population structure of an endangered orchid ( Dendrobium loddigesii Rolfe) from China revealed by SRAP markers. Scientia Horticulturae 129: 877-881.  Cameron, KM. Chase, MW. Whitten, WM. Kores, PJ. Jarrell, DC. Albert, VA. Yukawa, T. Hills, HG and Goldman, DH. 1999. A phylogenetic analysis of the Orchidaceae: evidence from rbcL nucleotide sequences. American Journal of Botany 86: 208-224.   CBOL Plant Working Group. 2009. A DNA barcode for land plants. Proceedings of the National Academy of Sciences, U.S.A 106: 12794-12797.
Chen, SL. Yao, H. Han, JP. Liu, C. Song, JY. Shi, LC. Zhu, YJ. Ma, XY. Gao, T. Pang, XH. Luo, K. Li, Y. Li, XW. Jia, XC. Lin, YL and Leon, C. 2010. Validation of the ITS2 region as a novel DNA barcode for identifying medicinal plant species. Plos One 5(1): e8613.  Ding, G. Li, X. Ding, X and Qian, L. 2009. Genetic diversity across natural populations of Dendrobium officinale, the endangered medicinal herb endemic to China, revealed by ISSR and RAPD markers. Russian Journal of Genetics 45: 327-334.  Fay, MF. Swensen, SM and Chase, MW. 1997. Taxonomic affinities of Medusagyne oppositifolia (Medusagynaceae). Kew Bulletin 52: 111-120.  Hollingsworth, PM. Graham, SW and Little, DP. 2011. Choosing and using a plant DNA barcode. Plos One 6(5): e19254.  Hou, B. Tian, M. Luo, J. Ji, Y. Xue, Q and Xiaoyu, D. 2012. Genetic diversity assessment and ex situ conservation strategy of the endangered Dendrobium officinale (Orchidaceae) using new trinucleotide microsatellite markers. Plant Systematics and Evolution 298: 1483-1491.  Infat, P. Hemant, KS. Saurabh, R. Udai, CP and Babbar, SB. 2012. DNA barcoding of endangered Indian Paphiopedilum species. Molecular Ecology Resources 12: 82-90.   Kim, HM. Oh, SH. Bh, GS. ari, . Kim, CS and Park, CW. 2014. DNA barcoding of Orchidaceae in Korea. Molecular Ecology Resources 14: 499-507.   Kimura, M. 1980. A simple method for estimating evolutionary rates of base substitutions through comparative studies of nucleotide-sequences. Journal of Molecular Evolution 16: 111-120.   Kress, WJ. Wurdack, KJ. Zimmer, EA. Weigt, LA and Janzen, DH. 2005. Use of DNA barcodes to identify flowering plants. Proceedings of the National Academy of Sciences, U.S.A 102: 8369-8374.  Lee, NS. 2007. Dendrobium Sw. In The genera of vascular plants of Korea. Park, CW (ed.), Academy Publishing Co, Seoul. 1370 pp.
Li, X. Ding, X. Chu, B. Zhou, Q. Ding, G and Gu, S. 2008. Genetic diversity analysis and conservation of the endangered Chinese endemic herb Dendrobium officinale Kimura et Migo (Orchidaceae) based on AFLP. Genetica 133: 159-166.   Linnaeus, C. 1753. Epidendrum. In Species plantarum Tomus 2. Holmia. Impensis Laurentii Salvii, 952-954.
Moritz, C and Cicero, C. 2004. DNA barcoding: promise and pitfalls. Plos Biology 2(10): e354.  Nielsen, R and Matz, M. 2006. Statistical approaches for DNA barcoding. Systematic Biology 55: 162-169.   Pang, XH. Song, JY. Zhu, YJ. Xu, HX. Huang, LF and Chen, SL. 2011. Applying plant DNA barcodes for Rosaceae species identification. Cladistics 27: 165-170.  Posada, D and Crandall, KA. 1998. MODELTEST: testing the model of DNA substitution. Bioinformatics 14: 817-818.   Sang, T. Crawford, DJ and Stuessy, TF. 1997. Chloroplast DNA phylogeny, reticulate evolution, and biogeography of Paeonia (Paeoniaceae). American Journal of Botany 84: 1120-1136.   Shi, LC. Zhang, J. Han, JP. Song, JY. Yao, H. Zhu, YJ. Li, JC. Wang, ZZ. Xiao, W. Lin, YL. Xie, CX. Qian, ZZ and Chen, SL. 2011. Testing the potential of proposed DNA barcodes for species identification of Zingiberaceae. Journal of Systematics and Evolution 49: 261-266.  Sun, Y. Sinner, DZ. Liang, GH and Hulbert, SH. 1994. Phylogenetic analysis of Sorghum and related taxa using internal transcribed spacers of nuclear ribosomal DNA. Theoretical and Applied Genetics 89: 26-32.   Swartz, O. 1799. Dianome Epidendri generis. Linn. Nova Acta Regiae Societatis Scientiarum Upsaliensis 6: 61-88.
Swofford, DL. 2002. PAUP: phylogenetic analysis using parsimony and other methods (ver. 4.0). Sinauer Associates, Sunderland, MA.
Takamiya, T. Wongsawad, P. Sathapattayanon, A. Tajima, N. Suzuki, S. Kitamura, S. Shioda, N. Handa, T. Kitanaka, S. Iijima, H and Yukawa, T. 2014. Molecular phylogenetics and character evolution of morphologically diverse groups, Dendrobium section Dendrobium and allies. AoB Plants 6: plu045.  Thompson, JD. Higgins, DG and Gibson, TJ. 1994. CLUSTAL W: improving the sensitivity of progressive multiple sequence alignment through sequence weighting, position specific gap penalties and weight matrix choice. Nucleic Acid Research 22: 4673-4680.  Trapnell, DW. Hamrick, JL and Giannasi, DE. 2004. Genetic variation and species boundaries in Calopogon (Orchidaceae). Systematic Botany 29: 308-315.  Tsai, CC. Peng, CI. Huang, SC. Huang, PL and Chou, CH. 2004. Determination of the genetic relationship of Dendrobium species (Orchidaceae) in Taiwan based on the sequence of the internal transcribed spacer of ribosomal DNA. Scientia Horticulturae 101: 315-325.  Valentini, A. Pompanon, F and Taberlet, P. 2009. DNA barcoding for ecologists. Trends in Ecology and Evolution 24: 110-117.   White, TJ. Bruns, T. Lee, S and Taylor, JW. 1990. Amplification and direct sequencing of fungal ribosomal RNA genes for phylogenetics. In PCR Protocols: A Guide to Methods and Applications. Innis, MA. Gelfand, DH. Sninsky, JJ. White, TJ (eds.), Academic Press, Inc, New York. 315-322.
Xiang, XG. Schuiteman, A. Li, DZ. Huang, WC. Chung, SW. Li, JW. Zhou, HL. Jin, WT. Lai, YJ. Li, ZY and Jin, XH. 2013. Molecular systematics of Dendrobium (Orchidaceae, Dendrobieae) from mainland Asia based on plastid and nuclear sequences. Molecular Phylogenetics and Evolution 69: 950-960.   Xu, Q. Zhang, GQ. Liu, ZJ and Luo, YB. 2014. Two new species of Dendrobium (Orchidaceae: Epidendroideae) from China: evidence from morphology and DNA. Phytotaxa 174: 129-143.  Yao, H. Song, JY. Liu, C. Luo, K. Han, JP. Li, Y. Pang, XH. Xu, HX. Zhu, YJ. Xiao, PG and Chen, SL. 2010. Use of ITS2 region as the universal DNA barcode for plants and animals. Plos One 5(10): e13102. 
|
|
















